(前回から続く)
前回はヒト細胞でBIR(break-induced replication)と呼ばれるHRの一形態が実際に働いていることを述べた。そこでBIRの様子を図示したので再び参照されたい。
超絶FISHによるBIRの検出
このBIRがテロメラーゼ非依存性テロメア維持(ALT)でも働いているかどうかが今回の話だ。まず昨年10月にGagosとHalazonetisグループがEMBO Reportsに出した短い論文から紹介する。
ここで彼らが課題として取り上げたのは、ALT細胞がBIRを使っているかどうかを明かにすることであった。この点を追求するために、彼らは特殊な、本当に特殊なFISHを編み出した。これは同じ染色体標本に三回連続でFISHを行い、各回のFISHの後に顕微鏡観察、撮影する。多数の同じ染色体について3枚の写真を撮って、後でそのFISHのシグナルのパターン変遷を把握する(注1)。
これを完全に理解することはかなり困難だが、シグナルの出方のパターンで、いわゆる"conservative replication"が起こったかどうかが判定できる。conservative replicationは通常のDNA複製(こちらは"semi-conservative replication")では見られず、BIRの証拠として捉えられる。conservative replicationでは、結果的に片方の染色分体では二本鎖DNAの両方ともが新しく複製され、逆の染色分体では両方とも複製されずに鋳型として用いられる(注2)。ここで用いたFISHでは200 bpを上回るサイズでのハイブリダイゼーションが起こらないとシグナルが識別できない。BIRの特徴の一つは長いサイズの複製なので、この方法で検出されるものはBIRの一つの要件を満たしている。解析の結果、U2OS(ALT細胞)ではBIRの特徴である"conservative replication"が高頻度で起こっていることがわかった(注3)。
前回紹介したHalazonetisグループによる哺乳動物細胞でのBIRを示した論文ではPOLD3がBIRに必要であることがわかっている。そこで今回のGagos、Halazonetis論文では、テロメア複製にもPOLD3が必要かどうかをsiRNA法により検討した。その結果、POLD3タンパクレベルを低下させるとconservative replicationの頻度が低下することがわかった。さらにもう一つのサブユニットであるPOLD4の低下も頻度を低下させることも判明した。POLD3は彼らが先行論文(2,014年サイエンス)で明らかにしたとおり、哺乳動物細胞におけるBIRに必要な因子であり、テロメアでもまたBIRが行われていることを強く支持している。
以上から、(1) ALT細胞のテロメア維持のためにBIRが働いていること、および (2) BIRがテロメアでBIRが起こるためには、数あるDNAポリメラーゼのうちPOLD3とPOLD4が必要とされると結論付けた。
テロメア内でのDNA断裂で誘導されるDNA複製
上の論文では超絶FISHを駆使することで、ALT細胞がそのテロメア複製にBIRを用いていることが示された。しかし何分にも”超絶”的な技法なので、この手法自体を用いた追試験が簡単に行われるとは思えない。
その点昨年11月にネイチャーに出たGreenbergグループの論文では、より一般的な実験手技が使われていて、かつ多角的なデータが揃えられている。こちらの研究では、主に制限酵素をテロメア上で発現させた。これによりテロメアに特異的にDNA二本鎖断裂(DSBs)を生じさせている。このDSBsに続くテロメアでのDNA複製の亢進や、関連タンパクのテロメアへの動員などをトレースしている。
最初に得た結果は、DSBsの誘導によりテロメアでのDNA複製が亢進したとするデータだ(注4)。こうしたDNA複製はALT細胞にのみ見られ、テロメラーゼ陽性細胞ではきわめて低いレベルでしか検出されなかった。このことは、ALT細胞におけるテロメアのDNA損傷の修復には”長いサイズ”のDNA複製が起こっていることを示している。
このDSBsに誘導されたDNA複製に必要な因子をsiRNA法で同定したところ、やはりPOLD3が同定された(注5)。これらの結果から、この現象はBIRであることが強く示唆された。さらにSMARD法(DNAファイバー上での免疫染色とFISH)により実際に長いサイズの複製がテロメアで起こっていることもトレースできた。これらのデータから、DSBsに誘導されたDNA複製はBIRであると結論づけられた。
POLD3は通常のDNA複製で必要なDNAポリメラーゼを構成するサブユニットである。分裂酵母(S. pombe)ではこのサブユニットは必須遺伝子ではない。このことは、ヒトの細胞でもPOLD3が欠損している状態で、長期間生存することが可能かもしれない。この考えのもとに、U2OS細胞(ALT細胞)でPOLD3を欠損した細胞株をCRISPR/Cas9法で取得することを試みた。得られた細胞クローンのうち、幾つかはPOLD3タンパクのレベルが低いことがわかった。しかしPOLD3タンパクを完全に欠損したクローンは取得できなかった。このことはヒト細胞ではPOLD3が細胞の生残に必須であることを強く示唆している。実際POLD3を低レベルで発現しているクローンは、すべて遺伝子内にin-frameでの塩基欠損ができていた。すなわちこれはタンパク活性的には部分欠損である。
さて表現系として最も重要なことはテロメア維持に対するPOLD3の意義である。上記クローンを25回分裂増殖させてDNAを抽出した。そのテロメア長をサザンブロット法にて調べたところ、その短縮が認められた。したがってPOLD3なくしてテロメアが維持できないことは明らかである。こうしたデータからPOLD3がALTに関与しており、それは実際にテロメラーゼ非依存性のテロメア維持に関与していると結論づけられたた。
以上のまとめ
以上の二つの論文の内容をまとめて理解すると、(1) ALTの細胞のテロメアでは長いサイズ(>200 bp)でのDNA複製が起こっている。(2) この複製では"conservative replication"である。これらはBIRの特徴である。(3) このBIRにはPOLD3が必要である。(4) 同じくPOLD4が必要である。(4) POLD3のタンパクレベルの低いクローンではテロメアの短縮が起こる。
とりあえず両論文の内容を要約するとこうなる。だから”ALTはBIRによる”のだ。これでよいのだろうか?
(続く)
(注1)この3回連続FISHはかなり特殊で、手技の持つ一般性が低く、適用範囲がかなり狭い範囲に限られるので、この詳細をここで述べるのは気がひける。しかし本質的にはこの手技の理解なくして結論を評価できないので一応下に原理を図示しておく。これは当該論文の図を参考にして作り直した。
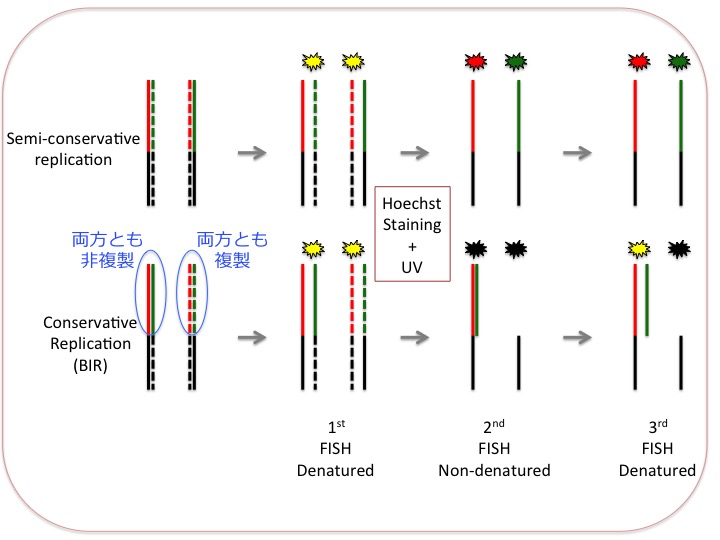
三段階のFISHを行って、同一染色体を各ステップごとに記録する。染色体標本作成に先立ってBrdUを培地に加えて新生鎖をラベルしておく。これは破線で示されている。図中赤、または緑で示したのはテロメア配列、黒はテロメアよりも近位の染色体配列。
最初のFISHは変性条件(二本鎖が乖離した状態)で行う。テロメア配列のG-rich鎖とC-rich鎖を別々の蛍光色素(赤と緑で示している)で標識したプローブを用いて検出する。全てのテロメア配列は赤と緑のシグナルの混合したもの、すなわち黄色となる。
2回目のFISHでは、以前書いたようにHoechst33258色素で染色した後、長波長UVで照射する。これによって新生鎖は傷んでハイブリダイゼーションできなくなる。FISH自体は染色分体をアニールさせた状態で行う。これによって鋳型鎖どうしは完全にハイブリするのでプローブで検出されなくなる(下段)。
3回目のFISHは再び変性条件で行う。これにより2段階で検出されなくなっていたシグナルが復活する。但し検出されるのは鋳型鎖のみだ。
このような込み入った手順で出てくるパターンの変遷を把握することにより、"semi-conservative"(上段)か"conservative"(下段、BIR)かの判定が可能となる。実際にはテロメアの一部が"conservative"になっているケースもあるがここでは省略した。手法の詳細については論文に当たられたい。
(注2)通常両方の鎖が複製されることは無論起こり得ない。これを理解するためには前回掲げた図を見て頂きたい。
(注3)この論文ではなぜかテロメラーゼ陽性細胞のデータとの比較が出されていない。ALT細胞とテロメラーゼ陽性細胞を比較することで明確な結論が得られるはずなので、これはこの論文の欠陥だ。
(注4)DSBsを誘導した後にU2OS細胞をBrdUでラベルして、回収された断片化DNAからBrdUを取り込んだ(すなわち新たに複製された)部分を回収する。これは抗BrdU抗体による免疫沈降で可能だ。これをドットブロット法で32Pでラベルされたテロメア配列をプローブとしたハイブリダイゼーションを行う。ブロット上の32Pを定量することでテロメアでのDNA複製を検出する。
(注5)この他にBIRのために必要であると特定された分子は、POLD1、RFC1、PCNAであった。一方、通常のDNA複製に必要なDNAポリメラーゼとしてはPOLEがあるが、これは不要であることがわかった。さらにHRに必要なRAD51、HOP2も不要であった。興味深いことだがテロメアBIRに必要な因子は、ふつうHRに要求される分子ではなく、一般のDNA複製に必要な因子であることが明らかとなった。